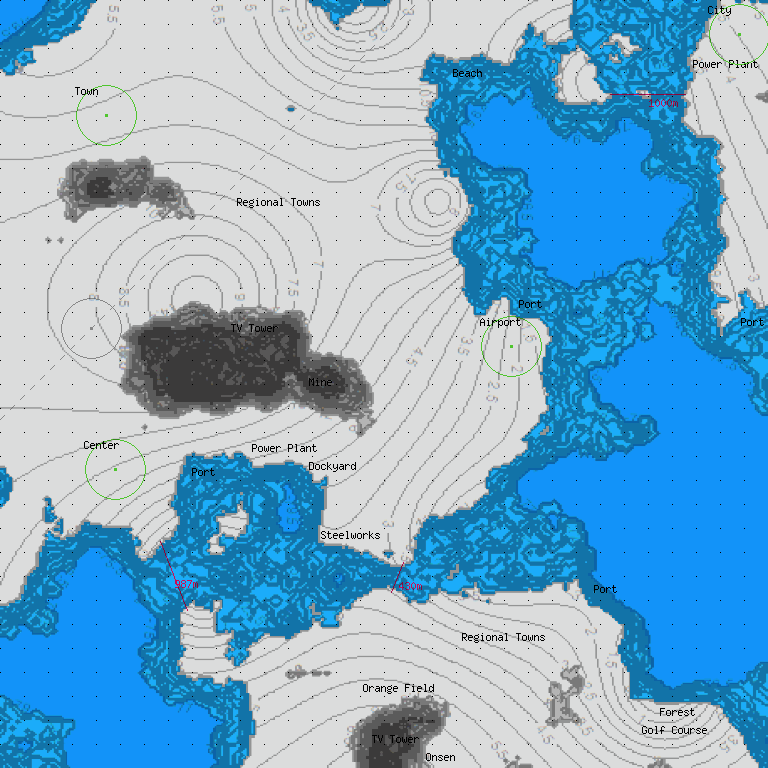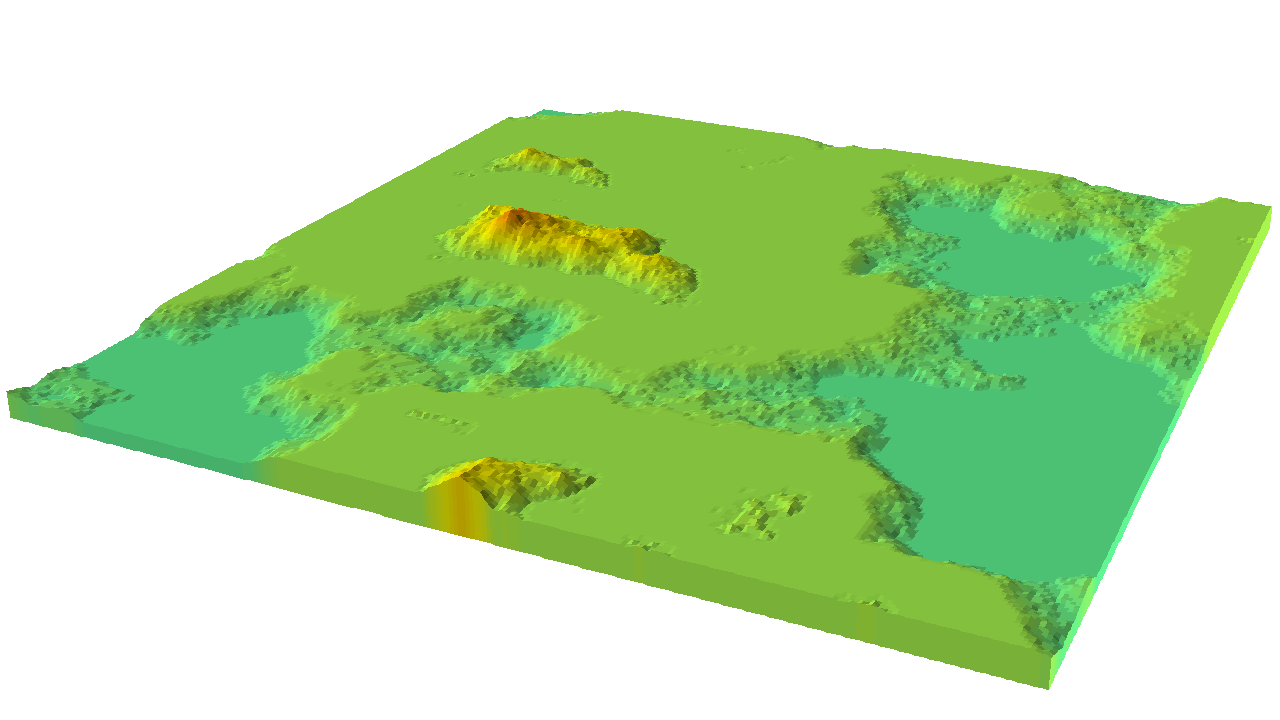
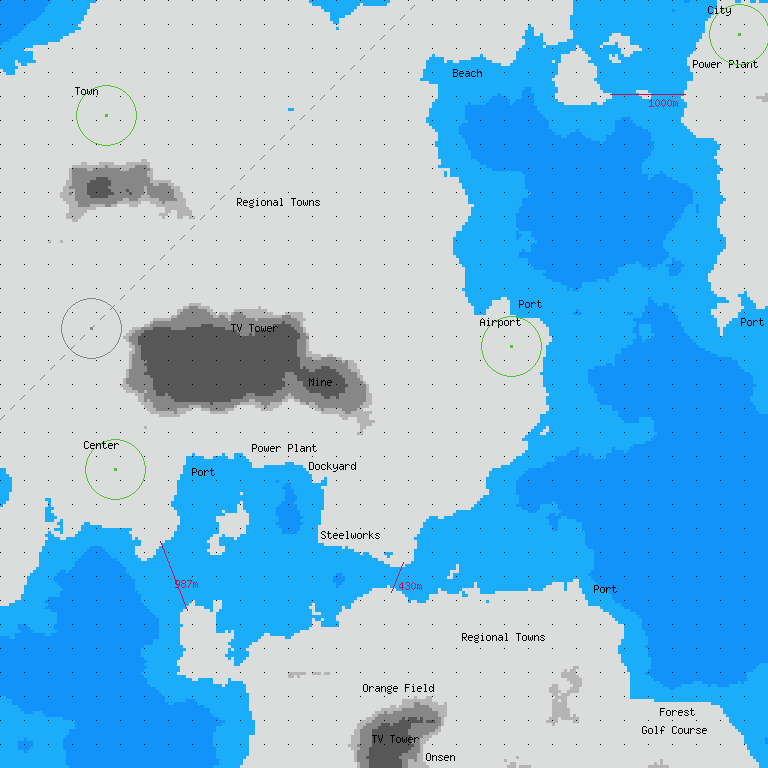
 (説明)
(説明)| サンプルデータ | 自動生成9号(x,y,z) |
|---|---|
| 自動生成9号(256x256) |
おもなシナリオマップについてはこちら。
| 「ベクトル円海山」のRスクリプト(k=9) |
|---|
| myakari3d0=read.table("clipboard",h=0) # XYZのほうのTSVデータ # TSVデータを読み込みます myakari3d <- rbind(subset(myakari3d0,V3> 0),subset(myakari3d0,V3< -3)) install.packages("mclust") # これは最初に1回だけ必要な操作です install.packages("ggvoronoi") # これは最初に1回だけ必要な操作です mycmpakari3d <- prcomp(myakari3d, scale=TRUE) mykmcmpakari3d <- kmeans(mycmpakari3d$x, 9, nstart=50) library(mclust) # 起動するたびに必要 clPairs(myakari3d, cl=mykmcmpakari3d$cluster) # プロットされた図を右クリックしてコピーや保存をします original <- mykmcmpakari3d$centers %*% t(mycmpakari3d$rotation) original <- scale(original, center = FALSE, scale = 1 / mycmpakari3d$scale) original <- scale(original, center = -mycmpakari3d$center, scale = FALSE) myakarioriginalcenters <- as.table(original) write.table(myakarioriginalcenters, file="clipboard", sep="\t") # クラスターの重心のリストをコピー myakari2d <- myakari3d[ ,c(1, 2)] plot(myakari2d, type="n") text(myakarioriginalcenters) rect(0, 0, 256, -256, border = "red") # プロットされた図を右クリックしてコピーや保存をします |
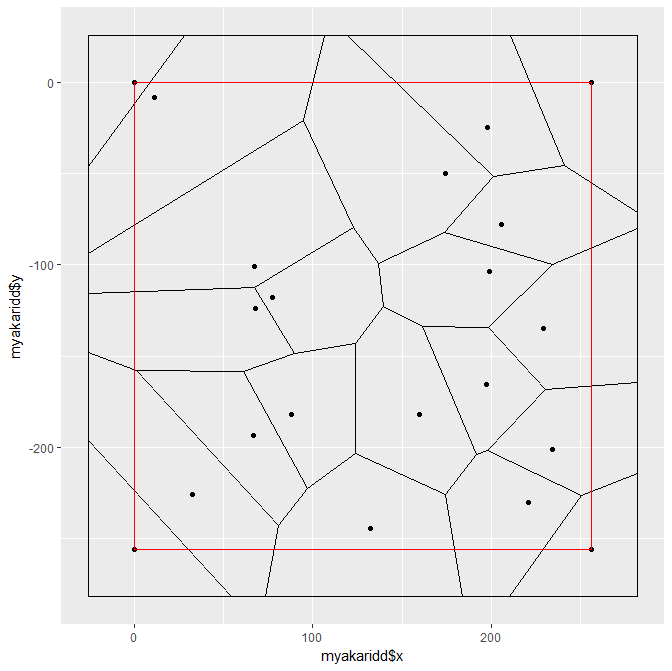
| ボロノイ図を描く |
| install.packages("ggvoronoi") # これは最初に1回だけ必要な操作です library(ggvoronoi) # 起動するたびに必要 myakaridd <- data.frame(original[,c(1,2)]) names(myakaridd) <- c("x", "y") myakaridd <- rbind(myakaridd, c(0,0), c(0,-256), c(256,0), c(256,-256)) # 4隅の点を追加 ggplot(myakaridd,aes(myakaridd$x, myakaridd$y)) + stat_voronoi(geom="path") + geom_point() + geom_rect(aes(xmin=0, xmax=256, ymin=-256, ymax=0), fill=NA, color="red") # プロットされた図を右クリックしてコピーや保存をします |
※クラスター数を変えて実行するときは青い行の「9」を書き換えます。
| 「ベクトル円海山」のRスクリプト | |
|---|---|
| (k=9) | (k=17) |
| myfunc1 <- function(a,b) sqrt(a ^ 2 + b ^ 2) myfunc2 <- function(delta,dist) ((delta * 2) * (1 / (dist ^ 2))) myfunc3 <- function(delta,max,min,height) ((delta - min) / (max - min)) * height myakari3dg <- rbind(subset(myakari3d0,V3 == 0),subset(myakari3d0,V3 == -3)) myakari2dg <- myakari3dg[ ,c(1, 2)] mydist1xg <- head(dist(rbind(myakarioriginalcenters[1, c(1,2)], myakari2dg), diag=FALSE, upper=FALSE), n=nrow(myakari2dg)) gc() mydist2xg <- head(dist(rbind(myakarioriginalcenters[2, c(1,2)], myakari2dg), diag=FALSE, upper=FALSE), n=nrow(myakari2dg)) gc() mydist3xg <- head(dist(rbind(myakarioriginalcenters[3, c(1,2)], myakari2dg), diag=FALSE, upper=FALSE), n=nrow(myakari2dg)) gc() mydist4xg <- head(dist(rbind(myakarioriginalcenters[4, c(1,2)], myakari2dg), diag=FALSE, upper=FALSE), n=nrow(myakari2dg)) gc() mydist5xg <- head(dist(rbind(myakarioriginalcenters[5, c(1,2)], myakari2dg), diag=FALSE, upper=FALSE), n=nrow(myakari2dg)) gc() mydist6xg <- head(dist(rbind(myakarioriginalcenters[6, c(1,2)], myakari2dg), diag=FALSE, upper=FALSE), n=nrow(myakari2dg)) gc() mydist7xg <- head(dist(rbind(myakarioriginalcenters[7, c(1,2)], myakari2dg), diag=FALSE, upper=FALSE), n=nrow(myakari2dg)) gc() mydist8xg <- head(dist(rbind(myakarioriginalcenters[8, c(1,2)], myakari2dg), diag=FALSE, upper=FALSE), n=nrow(myakari2dg)) gc() mydist9xg <- head(dist(rbind(myakarioriginalcenters[9, c(1,2)], myakari2dg), diag=FALSE, upper=FALSE), n=nrow(myakari2dg)) gc() # かなり時間がかかる場合があります mydeltaakari0 <- cbind(myfunc2(myakarioriginalcenters[1,3], myfunc1(mydist1xg, myakarioriginalcenters[1,3])), myfunc2(myakarioriginalcenters[2,3], myfunc1(mydist2xg, myakarioriginalcenters[2,3])), myfunc2(myakarioriginalcenters[3,3], myfunc1(mydist3xg, myakarioriginalcenters[3,3])), myfunc2(myakarioriginalcenters[4,3], myfunc1(mydist4xg, myakarioriginalcenters[4,3])), myfunc2(myakarioriginalcenters[5,3], myfunc1(mydist5xg, myakarioriginalcenters[5,3])), myfunc2(myakarioriginalcenters[6,3], myfunc1(mydist6xg, myakarioriginalcenters[6,3])), myfunc2(myakarioriginalcenters[7,3], myfunc1(mydist7xg, myakarioriginalcenters[7,3])), myfunc2(myakarioriginalcenters[8,3], myfunc1(mydist8xg, myakarioriginalcenters[8,3])), myfunc2(myakarioriginalcenters[9,3], myfunc1(mydist9xg, myakarioriginalcenters[9,3]))) # ここまでで1行です(改行を入れないでください) myakari0elv <- cbind(myakari2dg, myfunc3(rowSums(mydeltaakari0), max(rowSums(mydeltaakari0)), min(rowSums(mydeltaakari0)), 9.75)) colnames(myakari0elv) <- c("V1", "V2", "V3") mynewakari3d <- rbind(myakari3d, myakari0elv) |
mykmcmpakari3d <- kmeans(mycmpakari3d$x, 17, nstart=50) original <- mykmcmpakari3d$centers %*% t(mycmpakari3d$rotation) original <- scale(original, center = FALSE, scale = 1 / mycmpakari3d$scale) original <- scale(original, center = -mycmpakari3d$center, scale = FALSE) myakarioriginalcenters <- as.table(original) myfunc1 <- function(a,b) sqrt(a ^ 2 + b ^ 2) myfunc2 <- function(delta,dist) ((delta * 2) * (1 / (dist ^ 2))) myfunc3 <- function(delta,max,min,height) ((delta - min) / (max - min)) * height myakari3dg <- rbind(subset(myakari3d0,V3 == 0),subset(myakari3d0,V3 == -3)) myakari2dg <- myakari3dg[ ,c(1, 2)] mydist1xg <- head(dist(rbind(myakarioriginalcenters[1, c(1,2)], myakari2dg), diag=FALSE, upper=FALSE), n=nrow(myakari2dg)) gc() mydist2xg <- head(dist(rbind(myakarioriginalcenters[2, c(1,2)], myakari2dg), diag=FALSE, upper=FALSE), n=nrow(myakari2dg)) gc() mydist3xg <- head(dist(rbind(myakarioriginalcenters[3, c(1,2)], myakari2dg), diag=FALSE, upper=FALSE), n=nrow(myakari2dg)) gc() mydist4xg <- head(dist(rbind(myakarioriginalcenters[4, c(1,2)], myakari2dg), diag=FALSE, upper=FALSE), n=nrow(myakari2dg)) gc() mydist5xg <- head(dist(rbind(myakarioriginalcenters[5, c(1,2)], myakari2dg), diag=FALSE, upper=FALSE), n=nrow(myakari2dg)) gc() mydist6xg <- head(dist(rbind(myakarioriginalcenters[6, c(1,2)], myakari2dg), diag=FALSE, upper=FALSE), n=nrow(myakari2dg)) gc() mydist7xg <- head(dist(rbind(myakarioriginalcenters[7, c(1,2)], myakari2dg), diag=FALSE, upper=FALSE), n=nrow(myakari2dg)) gc() mydist8xg <- head(dist(rbind(myakarioriginalcenters[8, c(1,2)], myakari2dg), diag=FALSE, upper=FALSE), n=nrow(myakari2dg)) gc() mydist9xg <- head(dist(rbind(myakarioriginalcenters[9, c(1,2)], myakari2dg), diag=FALSE, upper=FALSE), n=nrow(myakari2dg)) gc() mydist10xg <- head(dist(rbind(myakarioriginalcenters[10, c(1,2)], myakari2dg), diag=FALSE, upper=FALSE), n=nrow(myakari2dg)) gc() mydist11xg <- head(dist(rbind(myakarioriginalcenters[11, c(1,2)], myakari2dg), diag=FALSE, upper=FALSE), n=nrow(myakari2dg)) gc() mydist12xg <- head(dist(rbind(myakarioriginalcenters[12, c(1,2)], myakari2dg), diag=FALSE, upper=FALSE), n=nrow(myakari2dg)) gc() mydist13xg <- head(dist(rbind(myakarioriginalcenters[13, c(1,2)], myakari2dg), diag=FALSE, upper=FALSE), n=nrow(myakari2dg)) gc() mydist14xg <- head(dist(rbind(myakarioriginalcenters[14, c(1,2)], myakari2dg), diag=FALSE, upper=FALSE), n=nrow(myakari2dg)) gc() mydist15xg <- head(dist(rbind(myakarioriginalcenters[15, c(1,2)], myakari2dg), diag=FALSE, upper=FALSE), n=nrow(myakari2dg)) gc() mydist16xg <- head(dist(rbind(myakarioriginalcenters[16, c(1,2)], myakari2dg), diag=FALSE, upper=FALSE), n=nrow(myakari2dg)) gc() mydist17xg <- head(dist(rbind(myakarioriginalcenters[17, c(1,2)], myakari2dg), diag=FALSE, upper=FALSE), n=nrow(myakari2dg)) gc() # かなり時間がかかる場合があります mydeltaakari0 <- cbind(myfunc2(myakarioriginalcenters[1,3], myfunc1(mydist1xg, myakarioriginalcenters[1,3])), myfunc2(myakarioriginalcenters[2,3], myfunc1(mydist2xg, myakarioriginalcenters[2,3])), myfunc2(myakarioriginalcenters[3,3], myfunc1(mydist3xg, myakarioriginalcenters[3,3])), myfunc2(myakarioriginalcenters[4,3], myfunc1(mydist4xg, myakarioriginalcenters[4,3])), myfunc2(myakarioriginalcenters[5,3], myfunc1(mydist5xg, myakarioriginalcenters[5,3])), myfunc2(myakarioriginalcenters[6,3], myfunc1(mydist6xg, myakarioriginalcenters[6,3])), myfunc2(myakarioriginalcenters[7,3], myfunc1(mydist7xg, myakarioriginalcenters[7,3])), myfunc2(myakarioriginalcenters[8,3], myfunc1(mydist8xg, myakarioriginalcenters[8,3])), myfunc2(myakarioriginalcenters[9,3], myfunc1(mydist9xg, myakarioriginalcenters[9,3])), myfunc2(myakarioriginalcenters[10,3], myfunc1(mydist10xg, myakarioriginalcenters[10,3])), myfunc2(myakarioriginalcenters[11,3], myfunc1(mydist11xg, myakarioriginalcenters[11,3])), myfunc2(myakarioriginalcenters[12,3], myfunc1(mydist12xg, myakarioriginalcenters[12,3])), myfunc2(myakarioriginalcenters[13,3], myfunc1(mydist13xg, myakarioriginalcenters[13,3])), myfunc2(myakarioriginalcenters[14,3], myfunc1(mydist14xg, myakarioriginalcenters[14,3])), myfunc2(myakarioriginalcenters[15,3], myfunc1(mydist15xg, myakarioriginalcenters[15,3])), myfunc2(myakarioriginalcenters[16,3], myfunc1(mydist16xg, myakarioriginalcenters[16,3])), myfunc2(myakarioriginalcenters[17,3], myfunc1(mydist17xg, myakarioriginalcenters[17,3]))) # ここまでで1行です(改行を入れないでください) myakari0elv <- cbind(myakari2dg, myfunc3(rowSums(mydeltaakari0), max(rowSums(mydeltaakari0)), min(rowSums(mydeltaakari0)), 9.75)) colnames(myakari0elv) <- c("V1", "V2", "V3") mynewakari3d <- rbind(myakari3d, myakari0elv) |
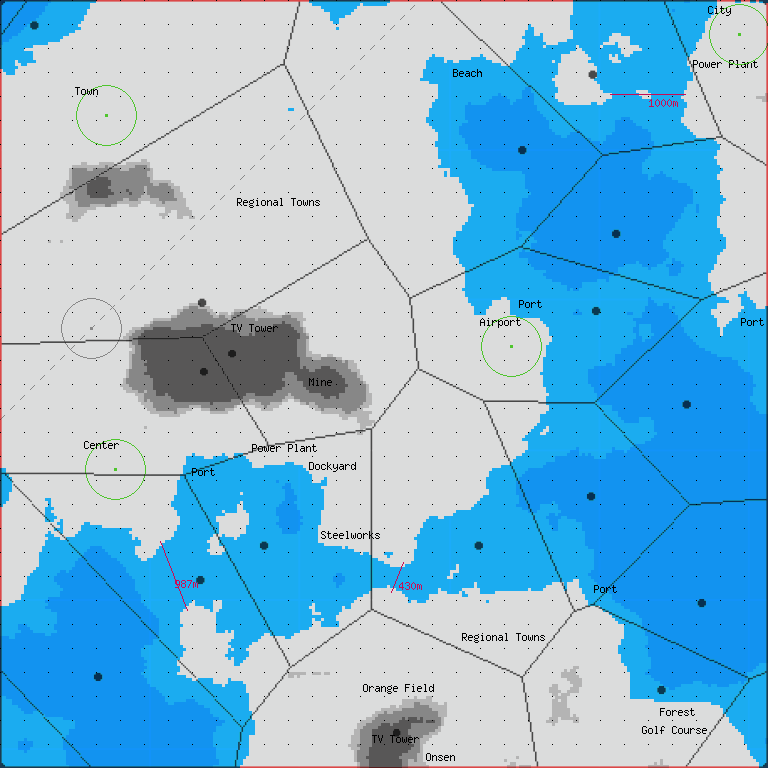
| 等高線を描く | |
| myakari=read.table("clipboard",h=0) # 256x256のほうのTSVデータ # TSVデータを読み込みます cpmyakari <- as.matrix(myakari) cpmyakari[(-1 * (myakari0elv$V2) + 1) + 256 * (myakari0elv$V1)] <- myakari0elv$V3 contour(cpmyakari, nlevels=1024) # プロットされた図を右クリックしてコピーや保存をします | |
(説明)